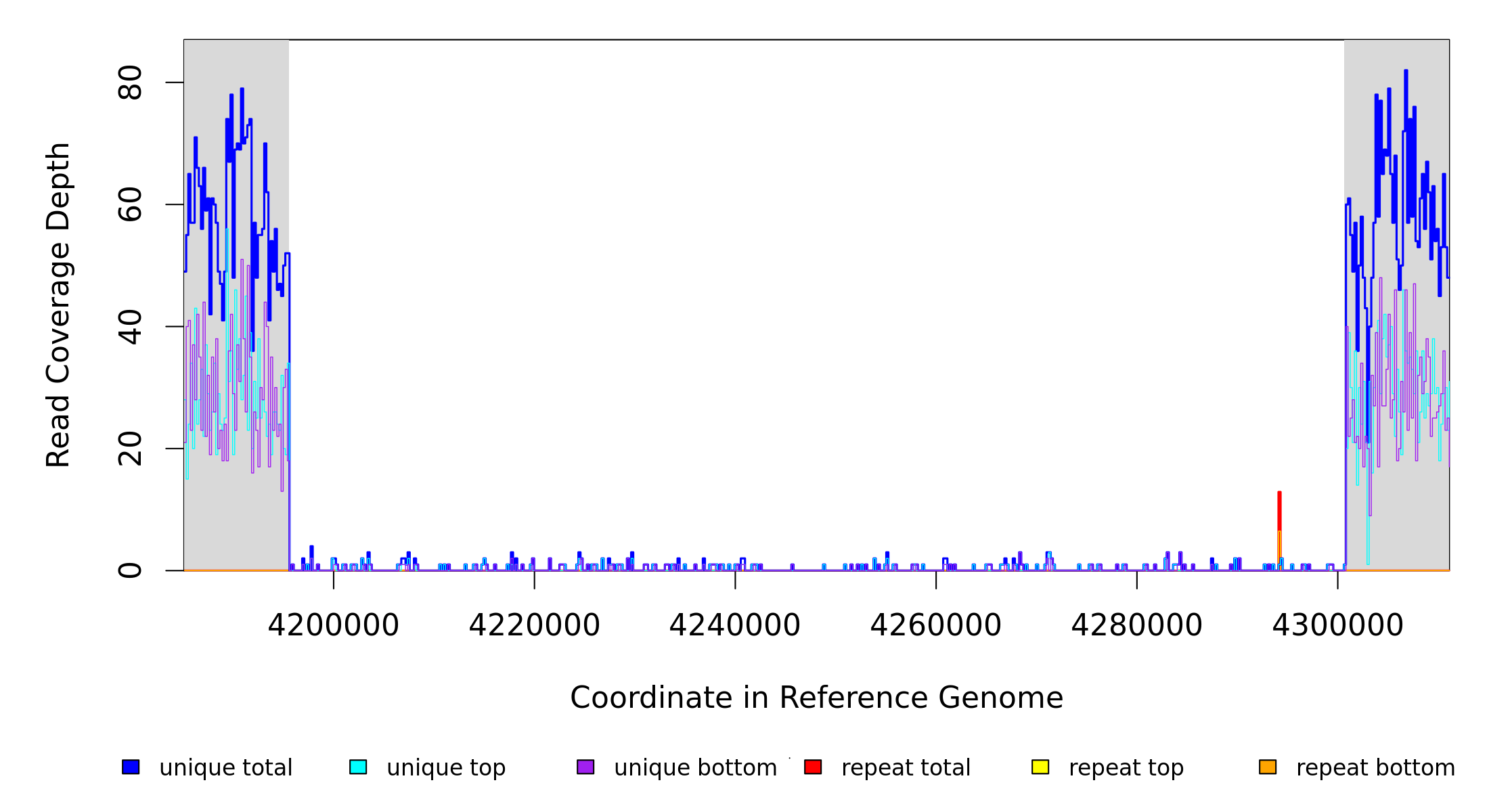
| MC JC |
ACB143 |
4,195,579 |
Δ105,043 bp |
|
[CJ019_03944]–[CJ019_04036] |
91 genes [CJ019_03944], CJ019_03945, CJ019_03946, CJ019_03947, CJ019_03948, CJ019_03949, CJ019_03950, CJ019_03951, CJ019_03952, CJ019_03953, CJ019_03954, CJ019_03955, CJ019_03956, CJ019_03957, CJ019_03958, CJ019_03959, CJ019_03960, CJ019_03961, CJ019_03962, CJ019_03963, CJ019_03964, CJ019_03965, CJ019_03966, CJ019_03967, CJ019_03968, catA_2, catC, catB, benM_3, CJ019_03973, spuC_4, spuD_3, kefF, aruH, alr, aruI_2, FCS1, acdA_1, CJ019_03982, rocE, rcsC_13, CJ019_03985, arcA_2, betI_2, CJ019_03988, CJ019_03989, CJ019_03990, CJ019_03991, CJ019_03994, nicP_10, pcaK_3, mrdA_1, CJ019_03998, CJ019_03999, glcC, CJ019_04001, CJ019_04002, lutA, CJ019_04004, CJ019_04005, gabR_1, yycB_2, CJ019_04008, CJ019_04009, bktB, hbd, fabR_1, cpdR, rcsC_14, cheB_3, cheR, rcsC_15, cckA_1, CJ019_04019, nicP_11, CJ019_04021, gloA, CJ019_04023, sdhL, CJ019_04025, CJ019_04026, CJ019_04027, CJ019_04028, CJ019_04029, CJ019_04030, soxA_2, rarD_3, prmC_3, proC_1, CJ019_04035, [CJ019_04036] |